
SNP检测服务是检测染色体基因组中单个核苷酸的突变而引起的DNA序列的多态性的技术服务,SNP以单个碱基的颠换、转换、插入和缺失等形式存在。作为第三代分子标记,SNP标记具有很大的发展潜力,被广泛应用于生物、农学、医学、生物进化等众多领域,在分子遗传学、药物遗传学、法医学以及疾病的诊断和治疗等方面发挥着重要作用。
SNP主要是指在基因组水平上由单个核苷酸的变异所引起的DNA序列多态性。与其它分子标记相比,SNP分辨率最高也最为丰富,覆盖基因组范围大,遗传上比较稳定,在人类基因组中,估计平均每1000个碱基对就有1个SNP,估计其总数可达300万个甚至更多,是继微卫星之后的第三代遗传标记。
SNP的分布不均匀,非转录序列中要多于转录序列,绝大多数位于蛋白的非编码区。通常情况下是一种二等位基因的变异,多为转换,即一种嘧啶碱基换为另一种嘧啶碱基,或一种嘌呤碱基换为另一种嘌呤碱基,转换与颠换之比为2:1。SNP在CG序列上出现最为频繁,而且多是C→T,因为CG中C即胞嘧啶常为甲基化的,自发脱氨后即变为胸腺嘧啶。
服务类型:
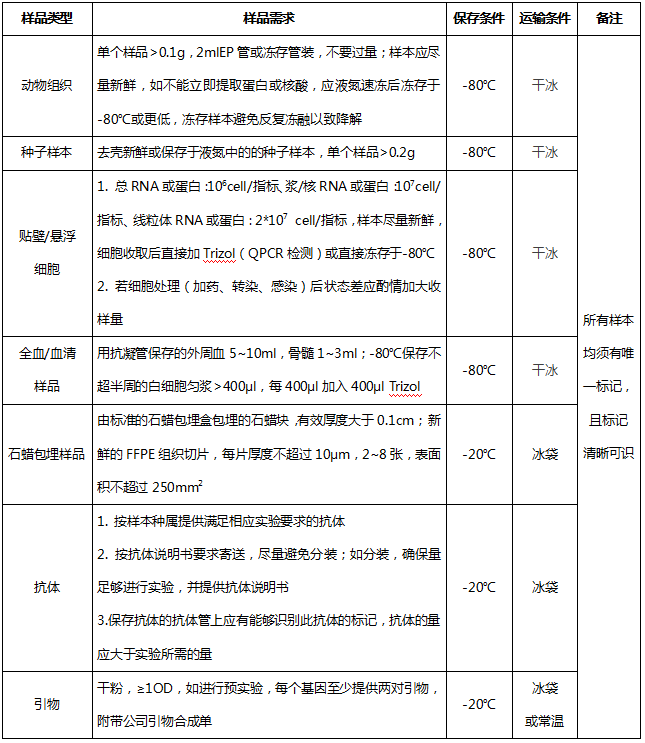
1.Taqman探针法(定性)
针对染色体上的不同SNP位点分别设计PCR引物和TaqMan探针,进行实时荧光PCR扩增。探针的5’-端和3’-端分别标记一个报告荧光基团和一个淬灭荧光基团。当溶液中存在PCR产物时,该探针与模板退火,即产生了适合于核酸外切酶活性的底物,从而将探针5’-端连接的荧光分子从探针上切割下来,破坏两荧光分子间的PRET,发出荧光。通常用于少量SNP位点分析。
高分辨率熔解曲线分析(HRM)是近几年兴起的SNP研究工具,它通过实时监测升温过程中双链DNA荧光染料与PCR扩增产物的结合情况,来判断是否存在SNP,而且不同SNP位点、是否是杂合子等都会影响熔解曲线的峰形,因此HRM分析能够有效区分不同SNP位点与不同基因型。这种检测方法不受突变碱基位点与类型的局限,无需序列特异性探针,在PCR结束后直接运行高分辨率熔解,即可完成对样品基因型的分析。该方法无需设计探针,操作简便、快速,成本低,结果准确,并且实现了真正的闭管操作。
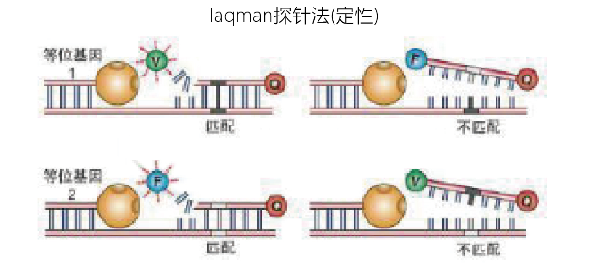
2.SNaPshot法测SNP
主要是基于荧光标记单碱基延伸原理的分型技术,也称小测序,主要针对中等通量的SNP分型项目。在一个含有测序酶、四种荧光标记ddNTP、紧临多态位点5’-端的不同长度延伸引物和PCR产物模板的反应体系中,引物延伸一个碱基即终止,经ABI测序仪检测后,根据峰的移动位置确定该延伸产物对应的SNP位点,根据峰的颜色可得知掺入的碱基种类,从而确定该样本的基因型。对于PCR产物模板可通过多重PCR反应体系来获得。该方法主要针对中等通量的SNP分型项目,通常用于10-30个SNP位点分析。
3.MassARRAy法(定量、多位点、高通量)
Sequenom公司的MassArray质谱系统在SNP分型市场上占有重要地位,是目前市场上拥有最高性价比的中高通量SNP分型检测系统,通过激光激发DNA分子片段,再用飞行时间来判断片段分子量的大小。该方法已经广泛地应用于遗传突变检测、SNP分型,是目前唯一采用质谱法进行直接检测的设备。MassARRAy法实验设计灵活,分型结果准确性高,性价比高。可对数百至数千份样本检测,样本通量从几百到几万。是中高通量SNP分型服务性价比最优的分型方法,适用于GWAS实验后续验证以及关联分析。
4.Illumina BeadXpress法
采用Illumina公司的BeadXpress系统进行批量SNP位点检测,可以同时检测1-384个SNP位点,往往用于基因组芯片结果确认,适合高通量检测。微珠芯片具有高密度、高重复性、高灵敏度、低上样量、定制灵活等特点,极高的集成密度,从而获得极高的检测筛选速度,在高通量筛选时可显著降低成本。
5.直接测序法
在所有SNP的检测方法中,对待检测片段进行直接扩增、测序是最为准确的方法,也是SNP分析的金标准。纯合型SNP位点的测序峰为单一峰型,而杂合型SNP位点的测序峰为套峰,因而很容易将其区分开来。通过直接测序方法进行SNP检测的检出率接近100%。该技术主要通过直接测序的方法来确定位点的基因型,这种分型方法准确性最高,但费用大。如果需要检测的几个SNP位点正好位于一个测序单元内(长度小于700bp),则单个位点的分型费用可显著降低,这种分型技术不失为一种良好选择。对于准确性要求特别高或样本量特别小(几个或几十个)的项目,这种分型技术很适合。
SNP可导致人类疾病,如癌症、传染性疾病、自身免疫性疾病等,SNP是研究人类家族和动植物品系遗传变异的重要依据,因此被广泛用于群体遗传学研究和疾病相关基因的研究,在药物基因组学、诊断学和生物医学研究中起重要作用。
应用领域:
1. 遗传图谱绘制的标记;
2. 疾病诊断和疾病易感基因的鉴别;
3. 药物基因组学研究和新药筛选;
4. 药物代谢和药物遗传学;
5. 法医鉴定和个体识别;
6. 群体遗传学研究和遗传多态性分析;
7. 物种鉴定、菌种鉴定等。
A. 直接测序法:
a、样品基因组DNA提取;
b、根据不同的区域进行引物设计、合成;
c、对所有样品进行基因扩增并纯化;
d、测序;
e、统计与分析。
B. TaqMan探针法:
a、样品基因组DNA提取与质检;
b、探针的设计、合成;
c、预实验优化PCR反应体系;
d、荧光定量PCR按测;
e、SNP位点数据分析。
C. SNaPshot法:
a、样品基因组DNA提取与质检;
b、引物的设计、合成;
c、扩增反应;
d、延伸反应;
e、测序仪检测;
f、数据分析
五、案例展示
Taqman探针法(定性)
SNaPshot法测SNP
六、 常见问题
① 为保证待测目的区域测序真实可靠,引物设计应该使待测目的区域边界距离上下游引物至少各50bp;
② 引物设计建议使用在线方式,以保证成功率;
③ 为保证测序敏感性,PCR产物片段大小应在250bp-650bp范围;
④ 为方便实验,建议引物合成时分装成1 管,方便将PCR与测序的引物分开;
⑤ 为保证引物的特异性,建议引物设计后在NCBI上blast确认;
⑥ 为防止降解,PCR产物应尽快测序,否则应该保存在-20℃,且时间不宜过长;
⑦ 为保证结果真实性,建议对关键点进行反向测序确认。
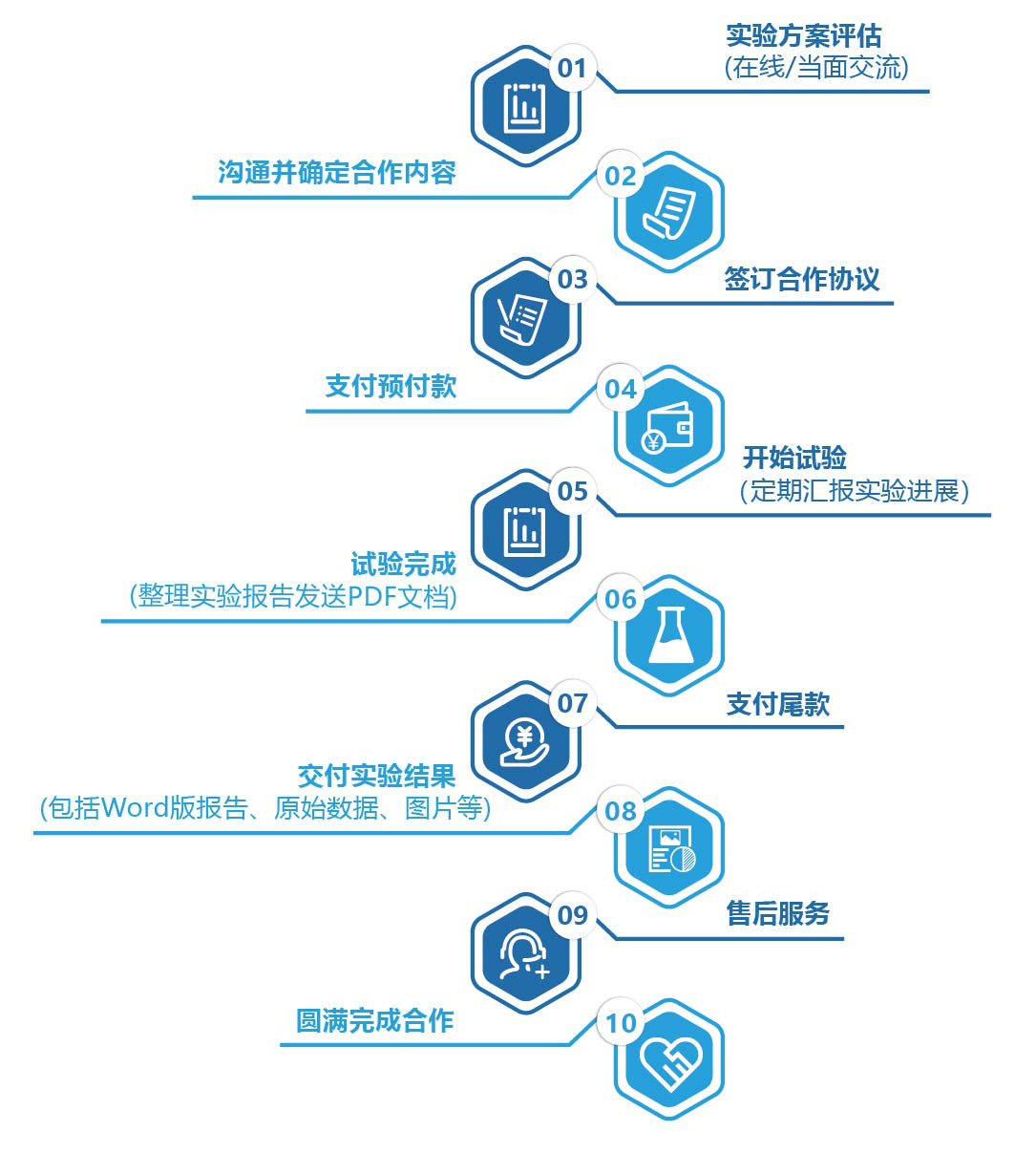
七、 服务流程
病理学检测
分子生物学检测
免疫学检测服务
细胞实验服务
动物实验平台